Molekulárněbiologická diagnostika vzácných mykóz stála dlouhou dobu v pozadí, ale v poslední době se začíná objevovat stále více prací, které skýtají do budoucna velkou naději. Tento článek je nabízí ve stručném přehledu.
Summary
Hrnčířová K., Lengerová M., Ráčil Z., Kocmanová I., Mayer J. Molecular diagnostics of invasive fungal infections caused by rare filamentous fungi
Number of invasive fungal infections caused by rare fungal pathogens (mainly zygomycetes) have increased in last years. Diagnostics of these infections is very complicated and application of specific detection of fungal DNA in clinical samples could be helpful. There is a stronger evidence of methods newly presented in last years that might be very promising for the future. This article offers a review of these methods.
Invazívní mykotické infekce jsou velmi závažnou, život ohrožující komplikací u imunokompromitovaných pacientů. Zatímco dříve mezi původci invazívních mykóz jasně dominovaly vláknité houby rodu Aspergillus a kvasinky rodu Candida, v posledních letech se stále častěji objevují infekce způsobené tzv. vzácnými mykotickými patogeny – zejména zygomycetami. Jedná se o ubikvitní organismy hojně přítomné zejména v půdě, prachu, stojící vodě a tlejících rostlinách, nejčastěji se tak do hostitelského organismu dostávají inhalací, ingescí, popřípadě kontaminací ran sporangiospórami z prostředí.(1)
Mezi vysoce rizikové skupiny pro rozvoj zygomykózy patří pacienti s diabetem, hematologickými a jinými malignitami, pacienti po orgánových transplantacích nebo po transplantaci krvetvorné tkáně, pacienti přetížení železem a pacienti léčení imunosupresívy, deferoxaminem nebo širokospektrálními antimikrobiálními léky.(2)
Původcem až 90 % všech zygomykóz je Rhizopus spp. (vůbec nejčastěji Rhizopus oryzae), dále pak Mucor spp., Absidia spp., Rhizomucor pusillus, Cunninghamella bertholletiae, Apophysomyces elegans a vzácněji některé další druhy. Kromě zygomycet však dochází k nárůstu výskytu i u dalších vláknitých hub – zejména Fusarium spp. a Scedosporium spp. a velmi vzácně se objevují také některé další (Acremonium spp., Alternaria spp., Paecilomyces spp., Scopulariopsis brevicaulis).(2–7)
Molekulárněbiologická diagnostika zygomykóz
Definitivní diagnóza v případě zygomykózy bývá zpravidla stanovena na základě histologického průkazu houbových vláken v postižené tkáni, kultivace pak umožňuje určení konkrétního druhu mykotického agens. Pro zygomycety jsou charakteristické široké, tenkostěnné, aseptované nebo velmi řídce septované hyfy větvící se v úhlu kolem 90°, často v prostředí zbytků rozrušené okolní tkáně.(8) I přes zřejmý histologický nález však může být kultivace ze vzorku tkáně negativní – kvůli předchozí antifungální léčbě, která u pacienta probíhala, popř. ztrátou životaschopnosti hyf v důsledku homogenizace vzorků tkáně.(9)
Zobrazovací metody (rtg, HRCT, CT) zpravidla neumožňují odlišení pozitivních nálezů od infekcí vyvolaných jinou vláknitou houbou. Vzhledem k celkové problematice diagnostiky těchto vzácných invazívních mykóz a naprosté absenci sérologických testů pro detekci zygomycet může být identifikace těchto patogenů v klinických vzorcích pomocí vysoce citlivých molekulárněbiologických metod velmi přínosná a v posledních letech se jejich vývoj těší velké pozornosti.
Výběr vhodného biologického materiálu
Podobně jako aspergilová PCR je i molekulárněbiologická detekce zygomycet velmi problematická a ani zde doposud nemáme k dispozici žádný standardizovaný postup. Buněčná stěna zygomycet, stejně jako ostatních vláknitých hub, je velmi odolná vůči rozrušení, houbové DNA bývá ve vzorcích velmi malé množství a i zde je problémem možnost kontaminace tzv. airborne fungi – tedy mikromycetami hojně přítomnými v okolním prostředí, a to v průběhu celého procesu od izolace DNA až po vlastní PCR detekci.
Při výběru vhodného materiálu hraje důležitou roli lokalizace infekce, která závisí především na predisponujících podmínkách – u pacientů s diabetickou ketoacidózou je nejčastější rinocerebrální forma, u neutropenie plicní nebo diseminovaná forma, u léčených kortikosteroidy plicní, diseminovaná a rinocerebrální, u léčených deferoxaminem diseminovaná forma a u pacientů po traumatu nebo pokud je vstupní bránou do organismu hostitele katétr nebo místo injekce, je to pak kožní nebo podkožní forma zygomykózy.
Pro zygomykózy je navíc charakteristická přítomnost rozsáhlé angioinvaze s výslednou žilní trombózou a nekrózou tkání. S angioinvazí je spojena také schopnost zygomycet diseminovat krevním řečištěm z původního místa infekce do dalších orgánů.(10) Je tedy velmi pravděpodobné, že by zygomycetová DNA mohla být při infekci přítomna nejen v postižených orgánech, ale také v krvi. U plicních forem zygomykózy je pro diagnostiku vhodným materiálem bronchoalveolární laváž.
Většina doposud publikovaných prací se věnuje převážně detekci zygomycet v postižené tkáni jako v materiálu, ve kterém při aktivní infekci očekáváme vysokou nálož zygomycetové DNA, popř. jsou metody prezentovány pro použití k identifikaci druhů vykultivovaných z klinických vzorků. V některých publikacích autoři vůbec neuvádí testování metod na klinických vzorcích, ale pouze na izolovaných kulturách,(11–14) což znemožňuje adekvátně zhodnotit jejich případnou vhodnost pro diagnostické použití.
Detekcí zygomycet ve vzorcích tkání (nativních nebo fixovaných v parafínu) se zabývá většina publikovaných prací, avšak pouze část z nich se může prezentovat větším množstvím otestovaných klinických vzorků.(15–19) Problém může nastat u tkáně fixované formalínem archivované v parafínových bločcích, kdy vlivem formalínové fixace může dojít k fragmentaci DNA ve vzorku, a tedy možným falešným negativitám při PCR amplifikaci.(20) Některé metody jsou také prezentovány jako detekce zygomycet v materiálu získaném ze zvířecích modelů invazívní zygomykózy,(21, 22) jedna práce na králičím modelu ukazuje také použití pro detekci zygomycetové DNA nejen ve tkáních, ale i ve vzorcích jiných materiálů – bronchoalveolární laváže (BAL) a plazmy.(23)
Izolace DNA
Izolace fungální DNA ze vzorku je klíčovým krokem pro molekulárněbiologickou diagnostiku. Zvolená metoda izolace musí být natolik účinná, aby dokázala rozrušit vysoce odolnou buněčnou stěnu mikromycet. Aby však byla použitelná pro rutinní diagnostiku, musí být zároveň jednoduchá a časově i finančně nenáročná. Zároveň je potřeba všemi dostupnými způsoby zamezit možnosti kontaminace vzorku mikromycetami z prostředí a samozřejmě také případné cross-kontaminaci mezi jednotlivými vzorky navzájem.
Bylo publikováno několik prací zabývajících se srovnáním účinnosti různých izolačních metod. Jejich výsledky naznačují, že velmi vhodným způsobem rozrušení fungálních buněk je mechanická lýza.(24–27) Také v publikacích týkajících se přímo detekce zygomycet je použití mechanické disrupce pomocí třepání se skleněnými či zirkoniovými kuličkami jako součásti izolačního procesu často používanou metodou,(13, 22, 28–30) oblíbené je také použití kitu pro izolaci DNA z tkání (DNeasy Tissue Kit, Qiagen), který obsahuje účinnější lyzační roztoky než běžné komerční kity, a to v kombinaci s mražením v tekutém dusíku a vařením,(15, 17, 18, 28) nebo inkubací vzorku v lyzačním roztoku přes noc v kombinaci s mechanickou disrupcí.(21)
S úspěchem je používán také automatický způsob izolace zygomycetové DNA s použitím instrumentu MagNA Lyser (Roche).(16, 23, 29) Na více než 80 vzorcích tkáně byla také prezentována metoda rozrušení buněk zygomycet pomocí 24hodinové inkubace v pufru obsahujícím 10% dodecylsulfát sodný (SDS) a proteinázu K.(19) Velkou nevýhodou této metody, stejně jako jiných metod využívajících rozrušení houbových buněk pomocí enzymů – ať už zmiňované proteinázy K, nebo lytikázy či zymolyázy, je však časová náročnost, kvůli níž nejsou tyto metody příliš vhodné pro rutinní diagnostické použití.
PCR – výběr vhodného úseku DNA
Obsah zygomycetové DNA v klinických materiálech bývá velmi nízký. Pro zvýšení citlivosti detekčních metod je tak velmi vhodné používat tzv. multikopiové geny. Vůbec nejčastěji používaným cílem jsou části genu pro ribosomální DNA (rDNA), který se v genomu zygomycet vyskytuje přibližně ve 100 kopiích(13) a skládá se ze tří konzervovaných podjednotek: 18S, 5,8S a 28S a variabilnějších oblastí ITS1 a ITS2 (internal transcribed spacer) mezi nimi – Obr. 1.
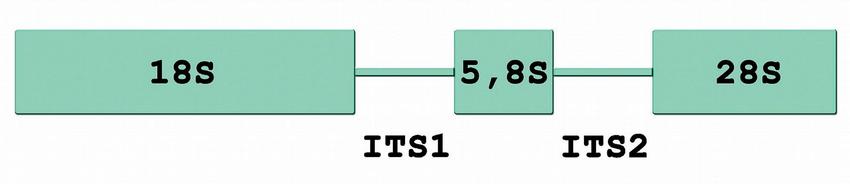
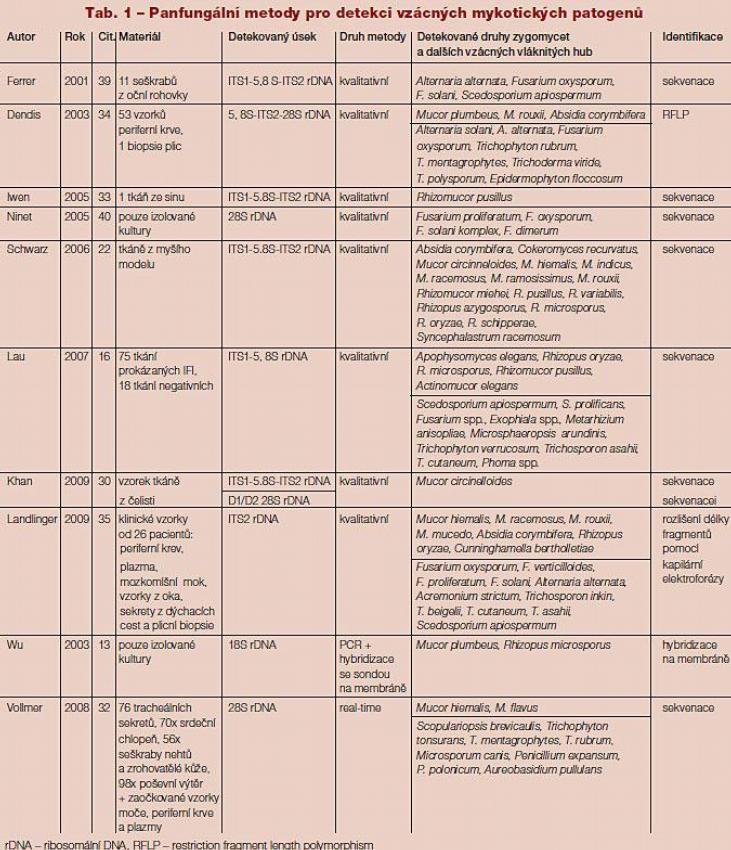
Pro jeho vysokou konzervovanost jsou části genu pro rDNA často využívány jako cíl panfungálních metod, které na jedné straně umožňují detekci velkého množství houbových druhů v jedné reakci, avšak použití nespecifických primerů s sebou zároveň přináší vyšší riziko detekce prostřeďových kontaminant (přehled publikovaných panfungálních metod použitých mimo jiné i pro detekci zygomycet včetně jednotlivých detekovaných druhů zobrazuje Tab. 1).
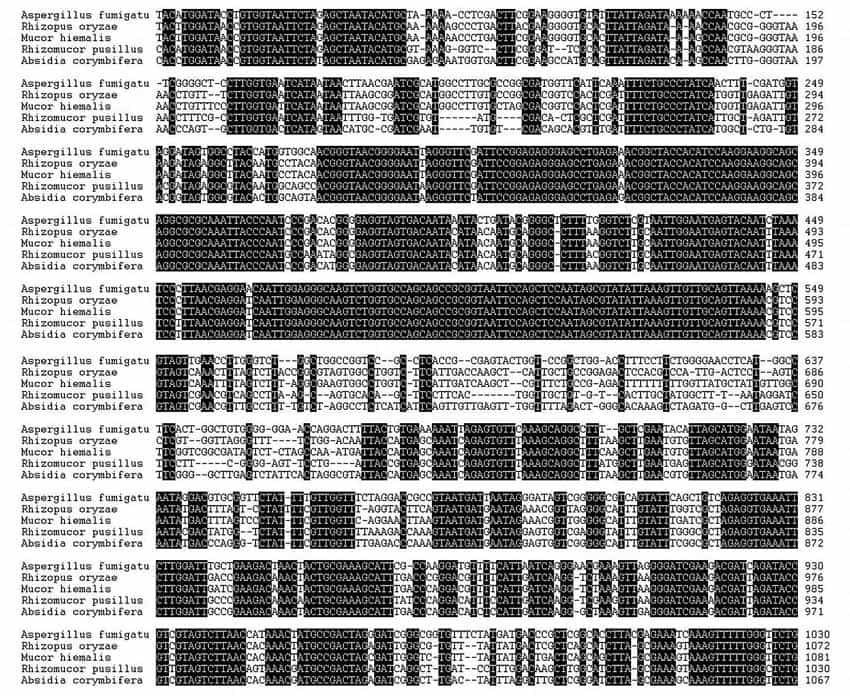
Velkou výhodou molekulární detekce zygomycet na rozdíl např. od Aspergillus spp. je však jejich sekvenční odlišnost od jiných vláknitých hub, které jsou ve vzorcích častými prostřeďovými kontaminantami (Penicillium spp., Cladosporium spp. aj.) Zygomytak lze specificky detekovat i v nejkonzervovanějším úseku rDNA – 18S, jak ukázali ve svých pracích Bialek a kol.(15, 17, 18) Na Obr. 2 je ukázka srovnání sekvencí části 18S rDNA některých klinicky významných zygomycet a Aspergillus fumigatus.
Více variabilní jsou úseky ITS1 a ITS2 rDNA. Bylo zjištěno, že v této oblasti se DNA některých zygomycet může lišit i v rámci různých kmenů jednoho druhu,(22, 31) což může komplikovat situaci při návrhu nových metod. U zygomycet byla na těchto úsecích ale pozorována také významná mezidruhová variabilita a díky tomu mohou být jednotlivé druhy odlišeny pomocí sekvenace,(16, 22, 30, 32, 33) což ale pro rutinní diagnostiku není z důvodu časové náročnosti příliš vhodné.
Další možností detekce produktů panfungálních metod je např. analýza délky restrikčních fragmentů (RFLP, restriction fragment length polymorphism)(34) nebo rozlišení přesné délky PCR produktů pomocí kapilární elektroforézy.(35)
Studie na zvířecích modelech
Použití zvířecích modelů je velmi přínosnou metodou pro studium infekčních onemocnění – jejich patogeneze, léčby, prevence a diagnostiky (Tab. 1). Nejčastěji se k těmto účelům používají laboratorní myši, králíci a morčata. Modelové infekce samozřejmě nelze zcela ztotožňovat s reálnou situací, zvířata bývají infikována velkým množstvím inokula, navíc kromě inhalační cesty, která je nejčastějším způsobem infekce u skutečných pacientů, jsou některé zvířecí modely infikovány intravenózně a průběh infekce tak může být zcela odlišný. Velkou výhodou je však to, že podmínky infekce mohou být snadno standardizovány.(36) V práci Ibrahim a kol.(21) autoři na diabetickém myším modelu studovali účinky antimykotika kaspofunginu.

Součástí studie byla také kvantifikace DNA R. oryzae specifickou real-time PCR metodou. Autoři pomocí této metody sledovali množství rhizopové DNA v různých časových obdobích a různých orgánech u myších modelů infikovaných nitrožilně různým množstvím inokula R. oryzae a uvádí, že tato metoda velmi dobře reflektovala míru rozvoje onemocnění. U neléčených jedinců 72 hodin po infekci bylo nejvyšší množství rhizopové DNA zaznamenáno v mozkové tkáni, nižší množství pak ve tkáni sekundárně zasažených orgánů – ledvinách, slezině, játrech a v plicích. Nejnižší množství inokula, po kterém byl po izolaci DNA z tkáně ještě zaznamenán pozitivní PCR signál, bylo 1x 104 spór.
Intravenózní způsob infekce myších modelů zygomykózy použili i Schwarz a kol.(22)
Infekce byla prováděna 6 různými druhy zygomycet – A. corymbifera, M. circinelloides, M. indicus, R. pusillus, R. microsporus var. rhizopodiformis a R. oryzae, v této studii však myši nebyly imunokompromitované a byly usmrceny třetí až čtvrtý den po infekci. Zygomycetová DNA byla poté detekována v mozkové a ledvinové tkáni pomocí panfungální PCR metody. Pozitivní histologický nález v obou tkáních mělo více než 50 % zvířat a u více než 70 % byl zachycen pozitivní PCR výsledek, což si autoři vysvětlují tím, že se ve tkáních mohly vyskytovat nevyklíčené spóry.
DNA ve vzorcích ze zvířecího modelu detekovali také Kasai a kol.(23) Svoji real-time PCR metodu prezenObr. nejdříve jako srovnání metod pro extrakci houbové DNA z kultur R. oryzae a zaočkovaných králičích BAL(29) a o několik měsíců později byla metoda publikována na 4 králičích modelech plicní zygomykózy vyvolané endotracheální infekcí imunosuprimovaných králíků druhy R. oryzae, R. microsporus, M. circinneloides, resp. C. bertholletiae. Jedná se o soubor dvou specifických real-time PCR, přičemž první detekuje rody Rhizopus, Mucor a Rhizomucor (qPCR-1, jednotlivé rody jsou rozlišovány na základě analýzy křivek tání) a druhá rod Cunninghamella (qPCR-2, druhy rozlišovány rovněž na základě analýzy křivek tání).
Autoři uvádějí, že metody nedetekují Candida spp., Aspergillus spp., Penicillium spp. apod. a ani lidskou nebo králičí DNA (bylo otestováno celkem 9 vzorků kvasinek a 21 vzorků vláknitých hub). Analytická senzitivita metod in vitro byla stanovena na 10 (qPCR-1) a 10–100 (qPCR-2) kopií plazmidové DNA na reakci, což odpovídalo 10 fg DNA R. oryzae, R. microsporus a M. circinelloides a 10 pg DNA R. pusillus.
Metodou qPCR-1 bylo otestováno 98 vzorků plicní tkáně a 22 BAL z experimentálního modelu, 97 (resp. 22) z nich bylo pozitivních. Metodou qPCR-2 bylo pozitivních všech 90 testovaných vzorků tkáně a 15 vzorků BAL. Senzitivita byla tedy stanovena na 99 % a 100 % (qPCR-1) a dvakrát 100 % (qPCR-2). DNA se podařilo detekovat také ve vzorcích plazmy – a to ve 20 z 50 (40 %) vzorků (qPCR-1), resp. 18 z 31 (58 %) vzorků (qPCR-2). Nevýhodou této metody však může být to, že nedetekuje klinicky významný rod Absidia.(23)
Studie specifických metod na klinických vzorcích
Pro zhodnocení použitelnosti publikovaných metod jsou velmi důležité výsledky jejich testování na klinických vzorcích. Na detekci Rhizopus spp., jako zygomycety nejčastěji izolované z klinického materiálu, je zaměřena kvalitativní metoda Nagao a kol. Autoři otestovali 5 vzorků od 3 pacientů s kožní zygomykózou – 1 vzorek kožní tkáně fixované v parafínu od každého pacienta a 2 vzorky séra od 1 pacienta. Ve dvou vzorcích tkáně byl identifikován R. oryzae a v jednom R. azygosporus, který byl zároveň identifikován i v obou vzorcích séra. V souběžně analyzovaném vzorku séra od zdravého dobrovolníka amplifikace žádného produktu neprobíhala.(28) Velmi přínosnou může být metoda specifická pro zygomycety publikovaná Bialek a kol.(15, 17, 18)
Jedná se o kvalitativní seminested PCR specifickou pro zygomycety řádu Mucorales. Rozlišení jednotlivých druhů bylo prováděno na základě sekvenace PCR produktů. Její detekční limit autoři uvádí 0,1 fg plazmidové DNA s vklonovaným PCR produktem. Pomocí této metody autoři ve vzorcích dokázali identifikovat následující druhy klinicky důležitých zygomycet: Rhizomucor spp. (v rámci tohoto úseku DNA od sebe není na základě sekvence možné odlišit druhy pusillus a miehei), M. hiemalis, A. corymbifera, R. oryzae, R. microsporus a Cunninghamella spp. V práci z roku 2007(18) nabízejí autoři srovnání výsledků histologické analýzy, kultivace a PCR z 56 vzorků tkáně imunokompromitovaných pacientů s podezřením na mykózu. Ze 6 histologických nálezů zygomycetových vláken bylo všech 6 vzorků PCR pozitivních, v jednom případě byla zjištěna souběžná infekce s A. fumigatus.
Kultivace byla pozitivní pouze ze 2 vzorků, autoři tak kladou důraz na to, že zvláště v případech, kdy kultivace selže, může být PCR s následnou sekvenací produktu velmi důležitým prostředkem pro určení konkrétního druhu zygomycety. Na konferenci ECCMID 2009 (European Congress of Clinical Microbiology and Infectious Diseases) byla publikována modifikace této metody, ve které bylo druhé kolo nahrazeno real-time PCR s následným přímým určením konkrétního druhu zygomycety pomocí High Resolution Melt (HRM) analýzy bez nutnosti následné sekvenace PCR produktů, což dokázalo významně zkrátit čas potřebný k analýze.( Real-time metodu pro detekci širšího spektra zygomycet představili Hata a kol.(19) (Tab. 2).
Metoda detekuje zástupce rodů Absidia, Apophysomyces, Cunninghamella, Mucor, Rhizopus a Saksenaea, jednotlivé rody jsou rozlišovány na základě analýzy křivek tání. Esej je hodnocena jako vysoce citlivá, umožňuje detekovat asi 10 kopií genu pro cytochrom b, zároveň nebyla zaznamenána cross-reaktivita s žádným z 59 testovaných nezygomycetových druhů. Rhizomucor spp., Conidiobolus spp. ani Syncephalastrum spp. metoda nedetekuje, ale do budoucna autoři plánují rozšířit detekované spektrum i o tyto druhy. V této práci bylo otestováno 81 vzorků tkání fixovaných v parafínu – 35 z nich bylo PCR a histologicky pozitivních, 19 bylo oběma metodami negativní a 27 bylo PCR negativních, ale histologicky pozitivních.
Výsledná senzitivita tedy byla 56,5 %. Toto je vysvětlováno možnou fragmentací DNA, a možnou inhibicí PCR reakce v důsledku použití formaldehydu ve fixovaných vzorcích. Testovány byly také 2 čerstvé tkáně s pozitivním histologickým nálezem a obě byly PCR pozitivní.
Zvláštní místo mezi metodami pro molekulární detekci mykotických patogenů zaujímá použití microarray – umožňuje totiž současnou specifickou detekci několika různých patogenů současně. Právě použití microarray by mohlo být v budoucnosti pro diagnostiku širokého spektra invazívních mykotických infekcí velkým přínosem. Metodu založenou na microarray publikovali Spiess a kol. v roce 2007.(38)
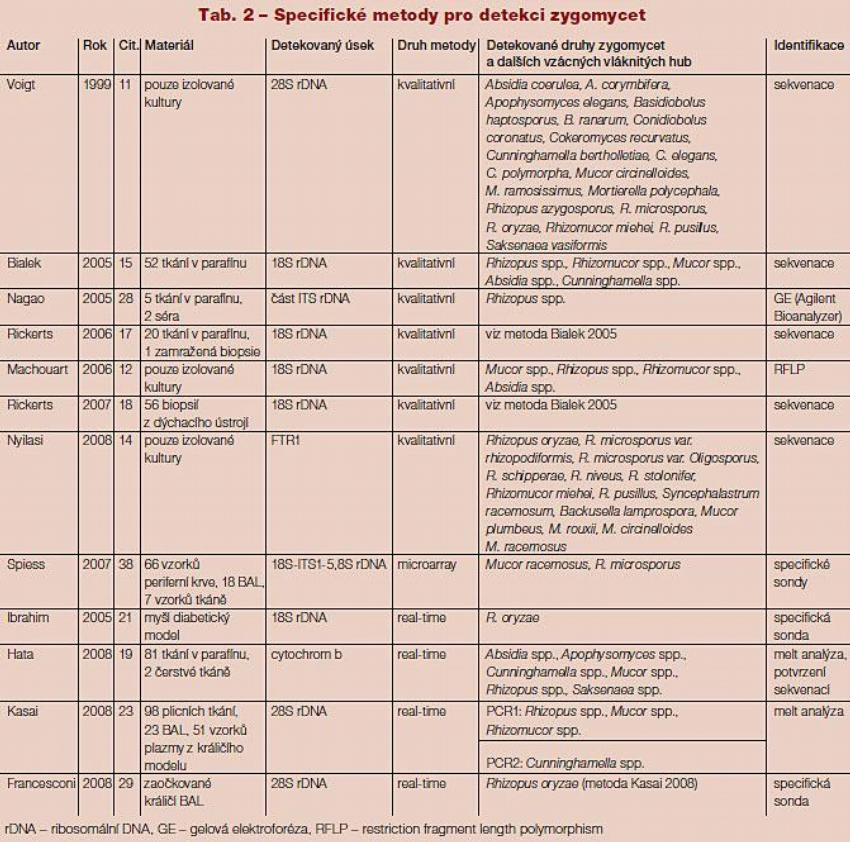
Kromě jiných fungálních patogenů metoda zahrnuje také detekci dvou klinicky významných zygomycet – R. microsporus a M. racemosus. Autoři uvádí detekční limit 300 pg (R. microsporus), resp. 10 pg (M. racemosus). Metodou byly otestovány vzorky krve, BAL a tkáně od 46 neutropenických imunokompromitovaných pacientů, R. microsporus byl identifikován v 1 vzorku krve. Autoři tuto metodu označují za vysoce specifickou, v 11 vzorcích však bylo detekováno dva a více různých patogenů současně (ve dvou případech i čtyři). Toto autoři vysvětlují možnou souběžnou infekcí pacienta několika různými druhy. Přehled všech zmíněných publikovaných metod pro specifickou detekci zygomycet zobrazuje Tab. 2.
Molekulárněbiologická diagnostika ostatních vzácných mykotických patogenů
Do této chvíle bylo publikováno pouze velmi malé množství prací týkajících se molekulární detekce ostatních vzácných mykotických patogenů. Často jsou k takové diagnostice používány panfungální metody s dodatečným určením konkrétního druhu pomocí sekvence,(16, 32, 39, 40) analýzy délky restrikčních fragmentů (RFLP)(34) nebo rozlišení přesné délky PCR produktů pomocí kapilární elektroforézy.(35) Některé z nich již byly představeny v předchozí části týkající se detekce zygomycet.
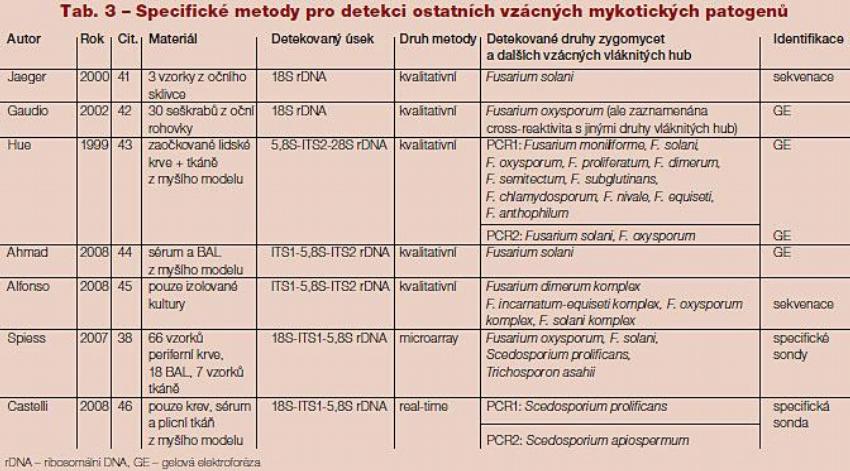
Další skupinu tvoří metody, které využívají panfungálního způsobu předamplifikace DNA v prvním kole nested PCR, následovanou amplifikací produktu s druhově specifickými primery v druhém kole.(41, 42) Kromě toho bylo publikováno také několik specifických PCR – kvalitativních pro Fusarium spp.(43–45) a microarray souběžně detekující Fusarium oxysporum, F. solani, Scedosporium prolificans a Trichosporon asahii.(38) V roce 2008 byla také publikována dvojice specifických real-time PCR pro detekci Scedosporium prolificans a apiospermum.(46) Publikované metody včetně spektra detekovaných druhů vzácných mykotických patogenů jsou shrnuty v Tab. 1 (panfungální metody) a Tab. 3 (specifické metody).
Závěr
Invazívní mykózy vyvolané tzv. vzácnými mykotickými patogeny se v posledních letech u imunokompromitovaných pacientů začínají objevovat stále častěji a je tedy logické, že i v oblasti molekulární diagnostiky jim začíná být věnována větší pozornost než dříve. Tato problematika je velmi komplikovaná, patogenů bývá ve vzorcích i při masívní infekci nízké množství a je zapotřebí velmi účinná metoda izolace fungální DNA. Zároveň je třeba dbát na zvýšené riziko možnosti kontaminace vzorků mikromycetami z prostředí. Velmi vhodné se proto zdá použití specifických metod, které tyto prostřeďové plísně nejsou schopny detekovat.
Kvantifikace nálože houbové DNA ve vzorku pomocí real-time PCR nám pak může pomoci odlišit případnou kontaminaci/kolonizaci od aktivní infekce. V posledních letech bylo publikováno několik metod pro specifickou detekci zygomycet a některých dalších vzácných mykotických patogenů, což by mohlo do budoucna vést k větší standardizaci jednotlivých postupů, zavedení panelů pro specifickou detekci více druhů mykotických patogenů a přispět tak ke zlepšení diagnostiky dříve raritních druhů invazívních mykotických infekcí.
Podpořeno grantem IGA MZ ČR NS10442-3/2009.
O autorovi: 1Mgr. Kristýna Hrnčířová, 1Mgr. Martina Lengerová, Ph. D., 1MUDr. Zdeněk Ráčil, Ph. D., 2Mgr. Iva Kocmanová, 1prof. MUDr. Jiří Mayer, CSc.
1Masarykova univerzita, Lékařská fakulta a Fakultní nemocnice Brno, Interní hematoonkologická klinika
2Fakultní nemocnice Brno, Oddělení klinické mikrobiologie
e-mail: khrncirova@fnbrno.cz

